
|
Archivos Latinoamericanos de Produccion Animal
Asociacion Latinoamericana de Produccion Animal
ISSN: 1022-1301 EISSN: 2075-8359
Vol. 14, Num. 4, 2006, pp. 154-160
|
Archivos Latinoamericanos de Produccion Animal, Vol. 14, No. 4, 2006, pp.
154-160
Evaluación genética de animales en poblaciones
multirraciales de bovinosutilizando modelos lineales
Genetic evaluation of animals in multibreed
cattle populationsusing linear models
M.A. Elzo1
Department of Animal Sciences, University of Florida, Gainesville, FL 32611-0910
Recibido Junio 28, 2006. Aceptado Agosto 10, 2006. 1E-mail: elzo@animal.ufl.edu
Code Number: la06027
ABSTRACT
This review discusses the use of linear procedures to evaluate cattle in multibreed populations for traits of economic importance. First, a multibreed mixed linear model is presented in detail, and all their terms are defined. Then, computational algorithms to solve multibreed mixed model equations and to estimate additive and nonadditive variance and covariance components are discussed, and the types of multibreed predictions obtained in these analyses are explained. Finally, applications of multibreed procedures to experimental and field datasets are presented, and implementations of national multibreed systems in the United States are discussed.
Key words: Cattle, Evaluation, Genetics, Linear, Model, Multibreed
RESUMEN
En esta revisión se discute la utilización de procedimientos lineales para la evaluación genética de bovinos en poblaciones multirraciales para caracteres de importancia económica. Primeramente, se presenta en detalle un modelo multirracial lineal mixto y se definen todos sus términos. A continuación se discute algoritmos computacionales para resolver las ecuaciones multirraciales de modelos mixtos y para estimar componentes de varianzas y covarianzas aditivas y no aditivas, y se explica los tipos de predicciones multirraciales que se obtienen. Finalmente, se presenta aplicaciones de procedimientos multirraciales con conjuntos de datos bovinos experimentales y a campo, y se discute implementaciones nacionales de sistemas multirraciales en Estados Unidos.
Palabras clave: Bovino, Evaluación, Genética, Lineal, Modelo, Multirracial
Introducción
Los procedimientos de evaluación genética de bovinos
han experimentado enormes cambios durante los últimos
30 años. Fundamental en este proceso fueron los trabajos
de investigación de C. R. Henderson. Henderson no solamente
desarrolló la metodología estadística de modelos lineales
mixtos para la evaluación genética de animales
(Henderson et al., 1959; Henderson, 1973, 1974), sino que
contribuyó con estrategias computacionales esenciales para
su aplicación a poblaciones grandes de animales. En particular,
Henderson desarrolló el algoritmo clásico que permitió calcular rápidamente
la inversa de la matriz de parentesco (Henderson, 1975; 1976). Posteriormente,
Quaas
(1976) desarrolló un algoritmo rápido y con pequeños requerimientos
de memoria para calcular la matriz de parentesco
en poblaciones con consanguinidad. Estos modelos
y algoritmos abrieron las puertas a la evaluación genética
de animales en poblaciones grandes con base a datos recolectados
a campo. Estos procedimientos fueron inicialmente
aplicados a poblaciones de ganado lechero. Aplicaciones a poblaciones de ganado
de carne tuvieron que esperar al
desarrollo de modelos que consideraran efectos propios
del animal (efectos directos) e influencias maternas (efectos
maternos). Tales modelos fueron desarrollados por
Quaas y Pollak (1980, 1981). Estos trabajos de investigación
marcaron el inicio del uso de modelos lineales mixtos
para evaluar animales pertenecientes a asociaciones de
ganado bovino de carne en Estados Unidos. Estos modelos
suponen que todos los animales en la población pertenecen
a la misma raza. Sin embargo, muchas razas se han
desarrollado utilizando cruzamientos entre animales de
varias razas o con cruzamiento absorbente hacia una raza
de tal manera que la población está realmente compuesta
de animales puros y cruzados. Estas poblaciones se denominan
poblaciones multirraciales. Elzo (1983) desarrollóuna metodología
lineal mixta para evaluar animales en poblaciones multirraciales dentro de un
país y a través de
países. Esta metodología incluyó efectos directos y maternos
aditivos y no aditivos, y heterogeneidad de varianzas
y covarianzas a través de combinaciones de grupos raciales.
Elzo también desarrolló algoritmos de inversión rápida de las matrices multirraciales de efectos genéticos aditivos (Elzo, 1990a) y no aditivos (Elzo, 1990b), y procedimientos para estimar componentes de varianza y covarianza (Elzo 1994, 1996). Arnold et al. (1992) describieron un modelo animal multirracial para ganado bovino de carne. Lo et al. (1993, 1995) presentaron un desarrollo algebraico más formal de modelos multirraciales. Procedimientos multirraciales para poblaciones grandes de ganado de carne fueron implementados por primera vez en la Universidad de Cornell en 1996 (Klei y Quaas, 1996; Klei et al., 1996) para evaluar animales puros y cruzados en la población multirracial Simmental-Simbrah-Canadian Simmental. En 2004, procedimientos multirraciales similares a los de la Universidad de Cornell fueron implementados en la Universidad de Georgia (Legarra et al., 2004). Ambas implementaciones multirraciales consideran solamente efectos no aditivos fijos (heterosis) y homogeneidad de varianzas y covarianzas a través de grupos raciales puros y cruzados. Procedimientos multirraciales desde el punto de vista bayesiano han sido desarrollados por Jara et al. (2001a,b), Cardoso y Tempelman (2004), y Cardoso et al. (2005).
En este trabajo se discutirán aspectos aplicados de la utilización de procedimientos lineales multirraciales para la evaluación genética de bovinos para caracteres de importancia económica con datos experimentales y a campo.
Modelo Multirracial Básico
Los elementos básicos de un modelo multirracial son similares a aquellos de un modelo unirracial que considere efectos aditivos y no aditivos. La mayor complejidad de los modelos multirraciales se debe a que tiene que explicar efectos ambientales, genéticos aditivos, y genéticos no aditivos en poblaciones compuestas de animales de varias razas puras y de animales cruzados de múltiples composiciones raciales. La manera como se explique cada uno de estos efectos y de las suposiciones asociadas con ellos determina la complejidad del modelo estadístico que se use. Una de las principales características de modelos multirraciales es que se permite la heterogeneidad de varianzas y covarianzas genéticas y ambientales a través de animales de cualquier combinación de razas parentales. Básicamente, un modelo multirracial para un carácter de producción contiene los siguientes efectos: 1) grupo contemporáneo multirracial, 2) otros efectos fijos (e.g., edad de la madre), 3) grupo genético aditivo, 4) grupo genético no aditivo, 5) efecto genético aditivo animal, 6) efecto genético no aditivo animal, y 7) residuo.
Grupo Contemporáneo Multirracial. Estos grupos están formados por animales de una raza o de múltiples composiciones raciales, de edades similares, del mismo sexo y sujetos a similares condiciones de manejo y alimentación. Tal como en evaluaciones unirraciales, grupos contemporáneos deben estar conectados a través del uso de padres en común. Estos padres pueden ser de razas puras o cruzados. Lo ideal sería realizar estas conexiones con padres de todos los grupos raciales representados en cada grupo contemporáneo para evitar problemas de interacciones entre grupos raciales de padres y grupos contemporáneos multirraciales.
Grupo Genético Aditivo Multirracial. Si se conocieran las relaciones de parentesco hasta los ancestros pertenecientes a las razas parentales de todos los animales en la población multirracial, los grupos genéticos aditivos se construirían en base a los animales con padres desconocidos de las razas parentales. Sin embargo, en muchos casos (e.g., poblaciones comerciales de ganado bovino de carne), esta información se desconoce. En tales casos, se utilizaría un sistema de regresión que proyectaría la información de aquellos animales puros y cruzados sin ancestros conocidos directamente hacia las razas parentales en función de su composición racial (Elzo, 1983; Elzo y Bradford, 1985; Elzo y Famula, 1985). Otra opción sería la creación de ancestros ficticios (Quaas, 1988; Westell et al., 1988) que servirían de intermediarios hacia los grupos raciales parentales. Esta opción requeriría de una suposición adicional: la composición racial de ancestros de animales cruzados que podría haberse obtenido de varias maneras.
Debido a que los efectos de las razas parentales no son estimables, los efectos genéticos aditivos raciales se estiman como desvíos de una base genética aditiva multirracial. Esta base genética multirracial puede estar compuesta de animales de una o más razas y grupos raciales nacidos (o que empezaron como reproductores) en un cierto año o dentro de un cierto período.
Grupo Genético No Aditivo. Los efectos no aditivos de grupo en poblaciones multirraciales se refieren a aquellos asociados con interacciones intrarraciales e interraciales dentro de un locus o en dos loci. Interacciones entre alelos de más de dos loci se suponen que son insignificantes. Estos efectos interraciales no aditivos se definen en función
de las probabilidades de tener alelos de diferentes razas en un locus o en
dos loci en progenies de toros de uno o varios grupos raciales apareados con
vacas de varios grupos raciales (Elzo, 1983, 1990b; Elzo y Famula, 1985; Elzo
y Wakeman, 1998). Debido a confusión de efectos en el modelo, los efectos interraciales intralocus se definen usualmente como desviaciones del promedio de los efectos intrarraciales intralocus. De manera similar, efectos interraciales entre alelos en dos loci se pueden definir como desvíos del promedio de las interacciones intrarraciales entre dos loci (Elzo, 1990b). Estos efectos genéticos intrarraciales intralocus y entre dos loci definen las bases genéticas no aditivas. Estimaciones de estos efectos no aditivos de grupo calculan el valor máximo
de las interacciones interraciales dentro de un locus o en dos loci.
Efecto Genético Aditivo Animal. Este efecto representa la suma de los efectos aditivos de todos los genes que afectan un carácter en un animal como desvío de la base genética aditiva multirracial (completo en el caso del modelo animal; la mitad en el caso de un modelo de padre y abuelo materno). Efectos genéticos aditivos multirraciales no se deben solamente a efectos genéticos aditivos intrarraciales sino también a efectos genéticos aditivos interraciales. Estos efectos genéticos aditivos interraciales son los llamados efectos de segregación (Wright, 1968; Lande, 1981; Lo et al., 1993). Predicciones de valores genéticos aditivos multirraciales permiten la comparación insesgada de animales de cualquier composición racial dentro de una población.
Efecto Genético No Aditivo Animal. Este efecto se puede calcular utilizando un modelo de subclase o uno de regresión (Elzo, 1990b). El modelo de regresión es más general, simple de calcular, y supone, al igual que los grupos genéticos no aditivos multirraciales, que las interacciones entre alelos en más de dos loci son insignificantes. El modelo de regresión predice los efectos intrarraciales e interraciales dentro de un locus o en dos loci en función de las probabilidades de tener alelos de una o más razas en un locus o en dos loci en progenies de un toro apareado con vacas de varios grupos raciales. Estos efectos aleatorios no aditivos se definen como desvíos de los grupos genéticos no aditivos para un locus y dos loci, y representan el valor máximo de un padre para cada efecto interracial. Si un padre, o alguno de sus parientes, tiene todas las razas de la población multirracial representadas en sus progenies, se puede predecir el valor genético no aditivo de progenies de este padre apareado con cualquier madre dentro de la población.
Supóngase que se quiere evaluar animales para peso al nacimiento (PN) y peso al destete (PD) en una población multirracial de bovinos compuesta de dos razas parentales (A y B) utilizando un modelo animal. En notación matricial, el modelo multirracial básico sería el siguiente:
y = Xb + Zgaga + Zgngn + Zaca + Zncn +v,
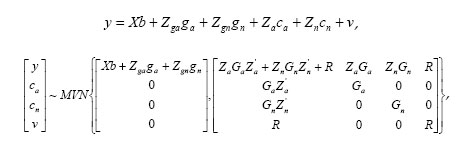
donde,
y = vector de registros de animales de PN y PD, ordenados por carácter dentro de animal,
b = vector de grupos contemporáneos multirraciales, y de otros efectos fijos (e.g., edad de la madre × sexo del ternero en función de la fracción A de la madre),
ga = vector de efectos de grupos genéticos aditivos directos y maternos intrarraciales (A) e interraciales (AB),
gn = vector de efectos de grupos genéticos no aditivos directos y maternos interraciales (A/B),
ca = vector de efectos genéticos aditivos multirraciales directos y maternos del animal c,
cn = vector de efectos genéticos no aditivos intrarraciales e interraciales directos y maternos para un locus y dos loci del animal c,
v = vector de residuos,
X = matriz que relaciona registros de animales a elementos del vector b,
Zga = matriz que relaciona registros de animales a elementos del vector ga a través de: 1) las probabilidades de tener alelos de la raza A en el animal para efectos genéticos directos intrarraciales, 2) las probabilidades de tener alelos de la raza A en la madre del animal para efectos genéticos maternos intrarraciales, 3) las probabilidades de tener alelos de las razas A y B en los padres del animal para efectos genéticos directos interraciales, y 4) las probabilidades de tener alelos de las razas A y B en los padres de la madre del animal para efectos genéticos maternos interraciales,
Zgn = matriz que relaciona registros de animales a elementos del vector gn a través de: 1) las probabilidades de tener alelos de las razas A y B en uno y dos loci del animal para efectos genéticos no aditivos interraciales directos, y 2) las probabilidades de tener alelos de las razas A y B en uno y dos loci de la madre del animal para efectos genéticos no aditivos interraciales maternos,
Za = matriz que relaciona registros de animales a elementos del vector ca: 1) al animal para efectos genéticos aditivos multirraciales directos, y 2) a la madre del animal para efectos genéticos aditivos multirraciales maternos,
Zn = matriz que relaciona registros de animales a elementos del vector cn a través de: 1) las probabilidades de tener alelos de una raza o de las dos razas en un locus o en dos loci del animal para efectos genéticos no aditivos directos, y 2) las probabilidades de tener alelos de una raza o de las dos razas en un locus o en dos loci de la madre del animal para efectos genéticos no aditivos maternos,
Ga = matriz de varianzas y covarianzas genéticas multirraciales aditivas directas y maternas entre los elementos del vector ca. Matriz Ga = (I - 0.5P)-1Ba(I - 0.5P')-1, donde P = matriz triangular inferior que relaciona padres y madres a animales con registros, P' = transpuesta de P, y
Ba = matriz diagonal en bloque con elementos iguales a varianzas y covarianzas genéticas
aditivas multirraciales entre elementos del vector ca. Formulas de covarianzas
genéticas multirraciales aditivas para cualquier número
de razas se dan en Lo et al. (1993) y Elzo (1994, 1995), y
para dos razas en Elzo y Wakeman (1998). Varianzas y
covarianzas genéticas multirraciales aditivas son funciones
lineales de varianzas y covarianzas aditivas
intrarraciales e interraciales. Varianzas y covarianzas en Ba se calculan con las reglas especificadas por Elzo (1990a).
Gn = matriz de varianzas y covarianzas genéticas no aditivas intrarraciales e interraciales entre elementos del vector cn. Matriz Gn = (I - 0.5P)-1Bn(I - 0.5P')-1, donde Bn = matriz diagonal en bloque con elementos iguales a varianzas y covarianzas genéticas no aditivas directas y maternas intrarraciales e interraciales para un locus y dos loci entre elementos del vector cn. Elementos de Bn se pueden calcular con las reglas especificadas por Elzo (1990b).
R = matriz diagonal en bloque de varianzas y covarianzas residuales entre elementos del vector v. Los bloques son de tamaño nt × nt, donde nt = número de caracteres (i.e., 2 × 2 en el caso de PN y PD). Varianzas residuales contienen varianzas ambientales multirraciales y varianzas debidas a efectos genéticos aditivos y no aditivos no contenidos en el modelo. Lo mismo sucede con covarianzas residuales. Varianzas y covarianzas ambientales son funciones lineales de varianzas y covarianzas ambientales intrarraciales e interraciales. Formulas para calcular varianzas y covarianzas ambientales y residuales para cualquier número de razas se presentan en Elzo (1994, 1995) y para dos razas en Elzo y Wakeman (1998).
Aspectos Computacionales
Resolución de las Ecuaciones Lineales Mixtas Multirraciales. La implementación de las ecuaciones lineales mixtas multirraciales requiere calcular las inversas de las matrices de varianzas y covarianzas genéticas aditivas multirraciales (Ga) y no aditivas (Gn) de manera eficiente. Elzo desarrolló algoritmos computacionales para obtener directamente: 1) los elementos de la inversa de Ga (Elzo, 1983, 1990a), y 2) los elementos de la inversa de Gn (Elzo, 1990b). Debido a que los bloques de la matriz R de varianzas y covarianzas de efectos residuales son pequeños, sus inversas se obtienen por métodos de inversión directa (e.g., Press et al., 1989).
Sistemas de ecuaciones multirraciales que contengan efectos genéticos no aditivos modelados como regresión generan un mayor número de contribuciones al conjunto de ecuaciones que con efectos no aditivos de subclases que tengan valores de uno o cero. Esto sumado al hecho de que las varianzas y covarianzas en un modelo multirracial varían a través de combinaciones de grupos raciales hace que el porcentaje de elementos que no sean cero en un sistema multirracial de ecuaciones sea mucho mayor (e.g., 30%) que en análisis unirraciales (e.g., 5%). La mayor cantidad de elementos hace que la resolución de sistemas de ecuaciones de modelos mixtos multirraciales requiera más tiempo que los unirraciales. Aún así, el rápido aumento en la capacidad de memoria, el incremento en la velocidad de procesamiento de los computadores personales y estaciones de trabajo, y los bajos precios de estas máquinas han contribuido a que aspectos computacionales no sean un obstáculo para la utilización de sistemas multirraciales.
Estrategias computacionales para resolver sistemas de ecuaciones multirraciales son similares a las unirraciales. En el caso de conjuntos de ecuaciones pequeños se resolverían utilizando métodos directos para resolver sistemas de ecuaciones (Golub y Van Loan, 1989; Press et al., 1989). Si su resolución fuera un paso intermedio en la estimación de varianzas y covarianzas, y se necesitara la inversa de la matriz del lado izquierdo de las ecuaciones, primero se obtendría esta inversa a través de procedimientos para matrices ralas (FSPAK, Perez-Enciso et al., 1994), y posteriormente se multiplicaría por el lado derecho. Si el número de ecuaciones o el número de elementos que no son cero fuera mayor que la capacidad de computación disponible se utilizaría métodos iterativos tales como Gauss-Seidel en bloque con sobre-relajación sucesiva (Golub y Van Loan, 1989; Press et al., 1989) o iteración sobre los datos (Schaeffer y Kennedy, 1986).
Estimación de Varianzas y Covarianzas. La construcción de las ecuaciones multirraciales de modelos mixtos supone que todas las varianzas y covarianzas genéticas aditivas, no aditivas, y ambientales son conocidas. Esta suposición es necesaria para obtener medias de cuadrados mínimos generalizadas que estiman los efectos fijos, y los mejores predictores lineales insesgados que predicen los efectos aleatorios cuando se resuelven las ecuaciones de modelos mixtos multirraciales. Como estas varianzas y covarianzas son en realidad desconocidas en la población, se les reemplaza con estimaciones obtenidas con el método de máxima verosimilitud restringida (Harville, 1977) obtenidas de los datos (Gianola et al., 1986). Elzo (1994, 1996) desarrolló procedimientos de estimación de varianzas y covarianzas aditivas, no aditivas, y ambientales de máxima verosimilitud restringida. Estos procedimientos estiman primero los elementos de la descomposición de Cholesky de las matrices de varianzas y covarianzas genéticas aditivas y ambientales de las razas parentales e interraciales de segregación, y de las matrices de varianzas y covarianzas genéticas no aditivas intrarraciales e interraciales utilizando un algoritmo de esperanzamaximización generalizado (Dempster et al., 1977). Este paso se hace para asegurarse que las matrices de covarianzas estimadas son simétricas positivas definidas. Posteriormente las matrices genéticas aditivas, no aditivas, y ambientales básicas se obtienen a través de la multiplicación de las matrices de Cholesky por sus transpuestas. Las matrices genéticas no aditivas básicas se utilizan como tales para obtener la inversa de Gn. Las matrices genéticas multirraciales aditivas y ambientales que se necesitan para el cálculo de las inversas de Ga y R se obtienen a través de funciones lineales de las matrices básicas aditivas y ambientales respectivamente. Elzo (1994, 1996) implementó este procedimiento en FORTRAN en el programa MREMLEM.
Predicciones Genéticas Multirraciales
Se puede obtener tres tipos de predicciones genéticas multirraciales: aditivas, no aditivas, y totales (Elzo, 1983, 1995, 2000). Las predicciones genéticas multirraciales aditivas son predicciones del valor genético medio esperado de un animal y constituyen la versión multirracial de las usuales predicciones genéticas intrarraciales aditivas. Se pueden utilizar de manera independiente para comparar animales de cualquier composición racial dentro de la población. Las predicciones genéticas no aditivas se refieren a las interacciones intralocus e interlocus entre alelos provenientes del padre y de la madre de un animal. Estas interacciones se recrean en cada gameto. Por ello, las predicciones no aditivas no se deberían usar solas, sino en combinación con las aditivas, como predicciones genéticas totales (i.e., aditiva + no aditiva).
Predicciones genéticas multirraciales aditivas y totales son de utilidad tanto a los criadores puros como a los productores comerciales. Predicciones genéticas multirraciales aditivas les servirían para seleccionar progenitores que les permitieran progresar genéticamente en sus rebaños. Predicciones genéticas multirraciales totales permitirían identificar a aquellos progenitores cuyas progenies se esperaría que fueran superiores para la combinación de efectos genéticos aditivos y no aditivos.
Si se usaran predicciones genéticas aditivas y totales para seleccionar toros para un rebaño comercial, en un paso inicial se podría escoger un grupo de toros por sus predicciones genéticas multirraciales aditivas, y en un paso posterior se seleccionaría el número de toros requerido con base a sus mente el mismo. Estos resultados sugirieron que se debería predicciones genéticas totales. Este procedimiento protege-explorar la evaluación genética de animales para efectos ría el progreso genético aditivo del rebaño, y también se es-genéticos aditivos, no aditivos, y totales en poblaciones peraría tener algo de beneficio genético no aditivo. bovinas multirraciales grandes.
Aplicación de Procedimientos Multirraciales
Probablemente la mayoría de los animales bovinos de
carne y un gran porcentaje de bovinos lecheros que existen
en el mundo tienen algún grado de cruzamiento. Sin embargo,
los procedimientos de evaluación genética utilizados
en la mayoría de estas poblaciones son probablemente
unirraciales o multirraciales simples que consideran efectos
fijos no aditivos solamente y suponen que las varianzas
y covarianzas a través de combinaciones de grupos raciales
son iguales. Estructura desbalanceada de los datos,
ausencia de razas puras y de ciertos cruzamientos, demasiadas
razas en la población, poca información de ciertas
razas, y falta de programas que puedan utilizarse para
evaluaciones genéticas multirraciales son algunos de los
factores que podrían estar influyendo en esta situación.
Evaluación Multirracial de Animales con Datos Experimentales.
Por su tamaño limitado, rebaños experimentales
bovinos apropiados para evaluaciones multirraciales deben
estar compuestos de pocas razas parentales. Por ello,
el rebaño multirracial de la Universidad de Florida fue diseñado
con solamente dos razas parentales, Angus y
Brahman, cuyas progenies cruzadas representaran apropiadamente
a los animales presentes en el estado de Florida.
El sistema de cruzamiento en este rebaño es dialélico,
donde padres Angus (A), ¾ A ¼ B, ½ A ½ B, ¼ A ¾ B,
Brahman (B), y Brangus (5/8 A 3/8 B) se cruzan con madres
de todos los grupos raciales. Este diseño permitió revalidar
la metodología multirracial para predecir valores
genéticos y estimar componentes de varianza para caracteres
de crecimiento (Elzo y Wakeman, 1998) y de canal
(Elzo et al., 1998b).
El rebaño Angus-Brahman de la Universidad de Florida
fue explícitamente diseñado para estudiar poblaciones
multirraciales. Sin embargo, rebaños multirraciales que tengan
diseño dialélico incompleto también pueden ser utilizados
en investigaciones de evaluación genética
multirracial, particularmente si el número de toros usados
por año fue grande y si se usaron toros de las razas
parentales y toros cruzados. Esta fue la situación en dos
rebaños experimentales de bovinos de carne en Colombia,
y en ambos casos, aún cuando el número de cruzamientos
fue limitado, hubo suficiente información para obtener predicciones
genéticas aditivas, no aditivas, y totales (i.e.,
aditivas + no aditivas) y estimaciones de varianzas y
covarianzas genéticas aditivas, no aditivas intralocus, y
ambientales para caracteres de crecimiento. El primer caso
fue el del rebaño multirracial Romosinuano-Cebú de
Turipaná (Elzo et al., 1998a) y el segundo fue el del rebaño
multirracial Sanmartinero-Cebú de La Libertad (Elzo et al.,
2001).
Estas investigaciones con datos experimentales indicaron
que existió variabilidad genética no aditiva y que el
ordenamiento de los toros por sus predicciones genéticas
multirraciales aditivas, no aditivas, y totales no fue exactamente el mismo.
Estos resultados sugirieron que se debería
explorar la evaluación genética de animales para efectos
genéticos aditivos, no aditivos, y totales en poblaciones
bovinas multirraciales grandes.
Evaluación Multirracial de Animales con Datos a Campo. Evaluación
de animales en poblaciones multirraciales comerciales va a requerir de suposiciones
más extensas que
las utilizadas en los modelos para datos de rebaños xperimentales.
Características de poblaciones multirraciales comerciales
incluyen: 1) utilización de más de dos razas
parentales, 2) apareamientos diseñados para obtener animales
de un número pequeño de composiciones raciales
específicas, y muchas veces el objetivo es el de obtener animales
de una sola composición racial cruzada (e.g., cruzamiento
absorbente, obtención de animales de 5/8 de una
raza y 3/8 de otra raza), 3) mayor número de grupos raciales
de padres que de madres (e.g., toros primordialmente de razas
puras, y vacas de cualquier composición racial de las
razas parentales consideradas que tengan buenas características
reproductivas, de salud, y maternas), 4) utilización
de múltiples toros para el apareamiento de vacas en monta
natural, 5) uso limitado de inseminación artificial, 6) uso
limitado de toros cruzados (inseminación artificial o monta
natural), y por ende, menor, y 7) uso de toros cruzados con
poca o ninguna información propia o de progenies. Debido
a estas características, los conjuntos de datos multirraciales
disponibles tienen una estructura extremadamente
desbalanceada, un gran número de razas parentales representadas
(muchas de ellas con escasa información), y con
un gran porcentaje de registros de producción y de pedigrícon información incompleta.
Información de poblaciones multirraciales a campo se
ha concentrado en organizaciones formadas por criadores
de bovinos en numerosos países. Estas organizaciones se
pueden clasificar en dos tipos: 1) asociaciones de criadores
de ganado bovino puro, y 2) organizaciones de productores
comerciales. Aunque el énfasis económico de estos
dos tipos de organización difieran, ambos forman parte de
la estructura genética y económica de la población
multirracial de la cual sus animales forman parte (e.g., criadores
puros proveen toros para productores comerciales).
En Estados Unidos muchas de las razas bovinas de carne
(e.g., Simmental, Gelbvieh) se originaron por cruzamiento
absorbente principalmente con vacas Hereford y Angus.
Por ello, estas asociaciones han mantenido un gran número
de registros de animales cruzados de diversas composiciones
raciales en sus bases de datos. En Brazil, además de
asociaciones de criadores de razas puras (ABCZ, 2006;
ANCP, 2006), existen organizaciones comerciales que reciben
datos de productores y conducen evaluaciones
genéticas (e.g., GENSYS Consultores Asociados, GENSYS,
2006).
Implementación de Evaluaciones Multirraciales Nacionales Implementación de evaluaciones nacionales siempre ha
involucrado compromisos entre exactitud de predicción
de valores genéticos y factibilidad de computación dada la
información disponible. Evaluaciones genéticas
multirraciales no son la excepción. Los dos sistemas de evaluación genética
multirracial para bovinos de carne en Estados Unidos: el de la Universidad de
Cornell y el de la
Universidad de Georgia son sistemas cuyos modelos estadísticos
tratan de extraer la mayor cantidad de información
genética aditiva y no aditiva promedio utilizando sistemas
computacionales apropiados para conjuntos de datos
grandes.
Sistema Multirracial de la Universidad de Cornell. El primer sistema nacional de evaluación genética multirracial en Estados Unidos fue desarrollado en la Universidad de Cornell en 1996 (Klei y Quaas, 1996; Klei et al., 1996). Su principal objetivo fue la evaluación simultánea de animales para efectos genéticos aditivos dentro de las poblaciones multirraciales Simmental y Simbrah (5/8 Simmental 3/8 Brahman) utilizando una metodología lineal mixta con varios componentes bayesianos. En particular, se utilizó metodología bayesiana para los efectos fijos de raza y de heterosis (Klei et al., 1996). La principal razón para la utilización de metodología bayesiana fue falta de información suficiente en el conjunto de datos disponible para la evaluación nacional. Además, debida a la falta de información en los datos y en la literatura, se crearon cuatro grupos de razas: Británicas (B; e.g., Angus, Hereford), Continentales (C; e.g., Simmental, Charolais), Zebu (Z; e.g., Brahman, Nelore), y Otras (O; e.g., Africander). Esta clasificación determinó 10 categorías de heterosis: B×B, B×C, B×Z, B×O, C×C, C×Z, C×O, Z×Z, Z×O, y O×O. Se supuso que la heterosis era proporcional a la probabilidad de tener alelos de diferentes razas en un locus, y que no había diferencias entre cruzamientos recíprocos. Aún cuando se incluyó heterosis en el modelo estadístico, las predicciones genéticas de los animales en la población continuaron siendo solamente de efectos genéticos aditivos. Este modelo genético multirracial es básicamente el que se utiliza actualmente en la Asociación Simmental Americana (ASA, 2006). Actualmente el catalogo de evaluación genética contiene predicciones genéticas para cuatro caracteres de crecimiento directo, cuatro caracteres maternos, y seis caracteres de canal.
Sistema Multirracial de la Universidad de Georgia. El segundo sistema multirracial en Estados Unidos fue desarrollado en la Universidad de Georgia en 2004 (Legarra et al., 2004). Este sistema se basó en el sistema implementado por la Universidad de Cornell y utilizó para su desarrollo el conjunto multirracial de datos de la asociación de criadores Gelbvieh. El sistema de agrupamiento de razas fue similar al de Cornell. Razas con representación suficiente de animales se mantuvieron como tales (e.g., Angus, Hereford, Shorthorn, Charolais), y otras razas con números pequeños de animales fueron agrupadas bajo las denominaciones de Británicas de carne, Británicas de leche, Continentales de carne, y Continentales de leche. Para la estimación de heterosis el agrupamiento fue Británica, Continental y Zebu, lo cual da seis categorías de heterosis (B×B, B×C, B×Z, C×C, C×Z, y Z×Z). El catálogo de Junio 2006 publicó predicciones genéticas de 16 caracteres reproductivos, de crecimiento y de canal (AGA, 2006). El modelo multirracial se utilizó solamente para los caracteres de crecimiento. Se utilizaron modelos unirraciales para el resto de los caracteres. Al igual que en la Universidad de Cornell, las predicciones genéticas obtenidas con el modelo multirracial en la Universidad de Georgia incluyen solamente efectos genéticos aditivos.
Consorcio Nacional de Evaluación de Bovinos de Carne. Este consorcio propuso una evaluación genética multirracial nacional en Estados Unidos (Garrick, 2004). Esta evaluación utilizaría información de todas las razas de carne en el país, lo cual es un tremendo desafío debido al tamaño de muchos de los conjuntos de datos y al grado de colaboración que se requiere entre asociaciones de criadores bovinos. Al igual que las evaluaciones multirraciales en las Universidades de Cornell y de Georgia, los procedimientos multirraciales utilizados por el consorcio aparentemente considerarían solamente efectos genéticos aditivos en las predicciones genéticas. En lugar de los caracteres usuales que se consideran en las evaluaciones genéticas nacionales, los animales se evaluarían por caracteres de relevancia económica. Caracteres de relevancia económica (Bourdon et al., 1997; Golden et al., 2000) dependen de objetivos económicos, los cuales pueden variar drásticamente a través de países y de regiones dentro de un país. Por ello, cualquier sistema que enfatice aspectos económicos deberá incluir un sistema que incluya: 1) reevaluación continua de los caracteres económicamente relevante que se estén utilizando, y 2) definición y evaluación de nuevos caracteres económicamente relevantes a medida que los actuales dejen de ser importantes.
Comentarios Finales
Evaluaciones genéticas de animales en poblaciones multirraciales bovinas han cambiado considerablemente durante la última década. Modelos utilizados en evaluaciones nacionales han aumentado notablemente en complejidad y algoritmos computacionales han aumentado en capacidad y sofisticación junto con tremendos avances en la capacidad de las máquinas de computación. Sin embargo, este es solamente el principio de un largo camino hacia la implementación de sistemas multirraciales cuyo objetivo sea la predicción del producto del apareamiento de animales individuales de diferentes razas o grupos raciales en términos de valores genéticos totales (i.e., aditivos + no aditivos) para caracteres de importancia económica. Para llegar allí se requerirá de una estrecha colaboración entre sectores comerciales y de investigación con el fin de obtener información suficiente para la implementación de modelos apropiados para cada población multirracial.
Literatura Citada
- ABCZ. 2006. Homepage. Available: http://www.abcz.com.br/.
- Accessed June 02, 2006. AGA. 2006. June 2006 Gelbvieh Sire Summary. American
Gelbvieh
- Association. Available: http://www.gelbvieh.org/
- breed_averages.htm. Accessed June 23, 2006.ANCP. 2006. Sire summaries.
Available: http://www.ancp.org.br/
- sumarios/index.asp. Accessed June 02, 2006.Arnold, J. W., J. K. Bertrand,
and L. L. Benyshek. 1992. Animal
- model for genetic evaluation of multibreed data. J. Anim. Sci.
- 70:3322-3332.
- ASA. 2006. 2006 Sire Summary - Spring Edition. American SimmentalAssociation.
Available: http://www.simmental.org/index.php?pageid=222. Accessed June 23,
2006.
- Bourdon, R., B. Golden, and B. Kinghorn. 1997. EPDs and beyond.Pages 74-87
in Proc. Beef Improvement Federation, Dickinson,ND.
- Cardoso, F. F., G. J. M. Rosa, and R. J. Tempelman. 2005. Multiplebreed
genetic inference using heavy-tailed structural models forheterogeneous residual
variances. J. Anim. Sci. 83:1766-1779.
- Cardoso, F. F., and R. J. Tempelman. 2004. Hierarchical Bayesmultiple-breed
inference with an application to genetic evaluationof a Nelore-Hereford population.
J. Anim. Sci. 82:1589-1601.
- Dempster, A. P., N. M. Laird, and D. B. Rubin. 1977. MaximumLikelihood
from incomplete data via the EM algorithm. J. RoyalStat. Soc., Ser. B, 38:1-38.
- Elzo, M. A. 1983. Multibreed sire evaluation within and across countries.
Ph. D. Dissertation, Univ. of California, Davis, CA.
- Elzo, M. A. 1990a. Recursive procedures to compute the inverse ofthe multiple
trait additive genetic covariance matrix in inbred andnoninbred multibreed
populations. J. Anim. Sci. 68:1215-1228.
- Elzo, M. A. 1990b. Covariances among sire by breed group of daminteraction
effects in multibreed sire evaluation procedures. J. Anim.Sci. 68:4079-4099.
- Elzo, M. A. 1994. Restricted maximum likelihood estimation of additive
and nonadditive genetic variances and covariances inmultibreed populations.
J. Anim. Sci. 72:3055-3065.
- Elzo, M. A. 1995. Considerations for the genetic evaluation ofstraightbred
and crossbred bulls in large multibreed populations.Pages 1-21 in Proc. Symp.
WRCC-100, Brainerd, MN.
- Elzo, M. A. 1996. Unconstrained procedures for the estimation ofpositive
definite covariance matrices using restricted maximumlikelihood in multibreed
populations. J. Anim. Sci. 74:317-328.
- Elzo, M. A. 2000. Multibreed Evaluation - Theory and Application.Pages
17-29 in Proc. 7th Genet. Pred. Workshop, Kansas City, MI.
- Elzo, M. A., and G. E. Bradford. 1985. Multibreed sire evaluation procedures
across countries. J. Anim. Sci. 60:953-963.
- Elzo, M. A., and T. R. Famula. 1985. Multibreed sire evaluation procedures
within a country. J. Anim. Sci. 60:942-952.
- Elzo, M. A., C. Manrique, G. Ossa, and O. Acosta. 1998a. Additiveand nonadditive
genetic variability for growth traits in theTuripaná Romosinuano-Zebu
multibreed herd. J. Anim. Sci.76:1539-1549.
- Elzo, M. A., G. Martinez, F. Gonzalez, and H. Huertas. 2001. Additive,
nonadditive, and total genetic variation and genetic predictionsfor growth
traits in the Sanmartinero-Zebu multibreed herd of LaLibertad. J. CORPOICA
3:123-139.
- Elzo, M. A., and D. L. Wakeman. 1998. Covariance components andprediction
for additive and nonadditive preweaning growth geneticeffects in an Angus-Brahman
multibreed herd. J. Anim. Sci.76:1290-1302.
- Elzo, M. A., R. L. West, D. D. Johnson, and D. L. Wakeman. 1998b. Genetic
variation and prediction of additive and nonadditive geneticeffects for six
carcass traits in an Angus-Brahman multibreedherd. J. Anim. Sci. 76:1810-1823.
- Garrick, D. 2004. Trends and developments in genetic evaluation ofbeef
cattle in the United States. Available: http://www.nbcec.org/nbcec/waf1.pdf.
Accessed June 23, 2006.
- GENSYS. 2006. Homepage. Available: http://www.gensys.com.br/. Accessed:
June 02, 2006.
- Gianola, F., J. L. Foulley, and R. L. Fernando. 1986. Prediction ofbreeding
values when variances are not known. Gen. Sel. Evol.18:485-498.
- Golden, B. L., D. J. Garrick, S. Newman, and R. M. Enns. 2000. A framework
for the next generation EPDs. Pages 2-13 in Proc. BeefImprovement Federation
32nd Annual Research Symposium andAnnual Meeting, Wichita, KS.
- Golub, G. H., and C. F. Van Loan. 1989. Matrix Computations. 2nd Ed.
The John Hopkins Univ. Press, Baltimore, MD.
- Elzo
- Harville, D. A. 1977. Maximum likelihood approaches to variancecomponent
estimation and to related problems. J. Am. Stat. Assoc.72:320-340.
- Henderson, C. R. 1973. Sire evaluation and genetic trends. Pages 1041 in
Proc. Anim. Breed. Symp. in Honor of Dr. J. L. Lush. ASAS-ADSA, Champaign,
IL.
- Henderson, C. R. 1974. General flexibility of linear model techniquesfor
sire evaluation. J. Dairy Sci. 57:963-972.
- Henderson, C. R. 1975. Rapid method for computing the inverse of arelationship
matrix. J. Dairy Sci. 58:1727-1730.
- Henderson, C. R. 1976. A simple method for computing the inverseof a numerator
relationship matrix used in prediction of breedingvalues. Biometrics 32:69-83.
- Henderson, C. R., O. Kempthorne, S. R. Searle, and C. M. von Krosigk.1959.
The estimation of environmental and genetic trends fromrecords subject to
culling. Biometrics 15:192-218.
- Jara, A., M. Elzo, y N. Barría. 2001a. Una aproximación bayesianapara la estimación de varianza en un modelo aditivoheterocedástico. I. Aspectos teóricos. Mem. XVII Reunión
ALPA,La Habana, Cuba. G36: 1964-1967.
- Jara, A., M. Elzo, y N. Barría. 2001b. Una aproximación bayesianapara la estimación de varianza en un modelo aditivoheterocedástico. II. Un estudio de simulación. Mem. XVII Reunión
ALPA,, La Habana, Cuba. G43:1936-1939.
- Klei, L., and R. L. Quaas. 1996. Multiple breed EPD: The Cornellapproach
to the Simmental data. Pages 41-49 in Proc. Symp.WCC-100 Regional Coordinating
Committee Meeting, Brainerd,MN.
- Klei, L., R. L. Quaas, E. J. Pollak, and B. E. Cunningham. 1996.Multiple-Breed
Evaluation. Pages 93-105 in Proc. 28th Res. Symp.and Annual Meeting,
Beef Improvement Federation, Birmingham,AL.
- Lande, R. 1981. The minimum number of genes contributing toquantitative
variation within and between populations. Genetics99:541-553.
- Legarra, A., J. K. Bertrand, T. Strabel, R. L. Sapp, and I. Misztal.2004.
Multi-breed genetic evaluation in a Gelbvieh population.Mimeograph, University
of Georgia, Athens, GA.
- Lo, L. L., R. L. Fernando, and M. Grossman. 1993. Genotypiccovariance between
relatives in multibreed populations: Additivemodel. Theor. Appl. Genet. 87:423-430.
- Lo, L. L., R. L. Fernando, and M. Grossman. 1995. Theory for modelingmeans
and covariances in a two-breed population with dominanceinheritance. Theor.
Appl. Genet. 90:49-62.
- Perez-Enciso, M., I. Misztal, and M. A. Elzo. 1994. FSPAK: An interface
for public domain sparse matrix subroutines. Proc. 5th World Congr.
Genet. Appl. Livest. Prod. 22:87-88.
- Press, W. H., B. P. Flannery, S. A. Teukolsky, and W. T. Vetterling.1989.
Numerical Recipes. The Art of Scientific Computing(FORTRAN Version). Cambridge
Univ. Press, NY.
- Quaas, R. L. 1976. Computing the diagonal elements and inverse ofa large
numerator relationship matrix. Biometrics 32: 949-953.Quaas, R. L. 1988.
Additive genetic model with groups and
- relationships. J. Dairy Sci. 71:1338-1345.
- Quaas, R. L. and E. J. Pollak. 1980. Mixed model methodology forfarm and
ranch beef cattle testing programs. J. Anim. Sci. 51:12771287.
- Quaas, R. L. and E. J. Pollak. 1981. Modified BLUP equations for siremodels
with groups. J. Dairy Sci. 64:1868-1872.
- Schaeffer, L. R. and B. W. Kennedy. 1986. Computing strategies forsolving
mixed model equations. J. Dairy Sci. 69:575-579.
- Westell, R. A., R. L. Quaas, and L. D. Van Vleck. 1988. Genetic groups
in an animal model. J. Dairy Sci. 71: 1310-1318.
- Wright, S. 1968. Evolution and the genetics of populations. Vol. 1.Genetics
and biometrical foundations. Univ. Chicago Press.
© 2006 ALPA. Arch. Latinoam. Prod. Anim.
|
